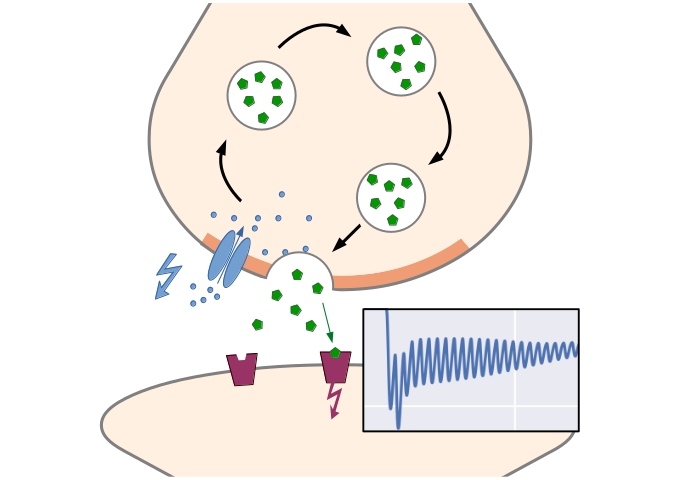
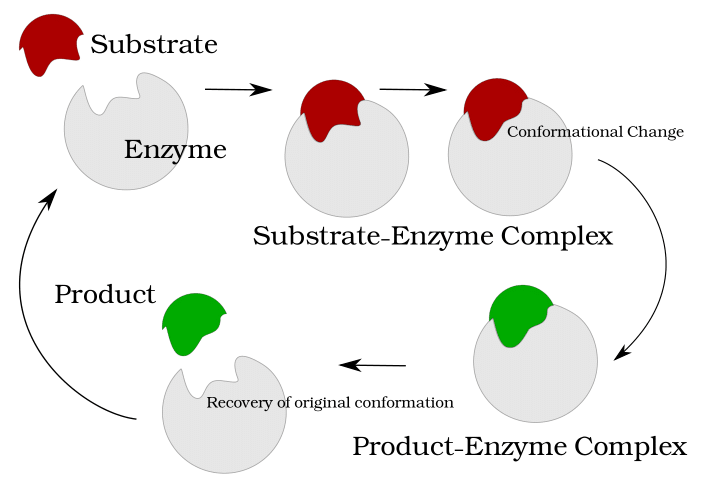
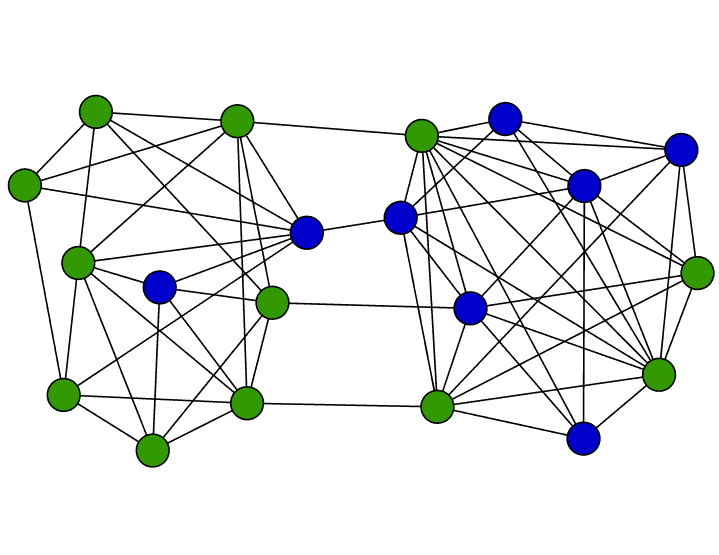
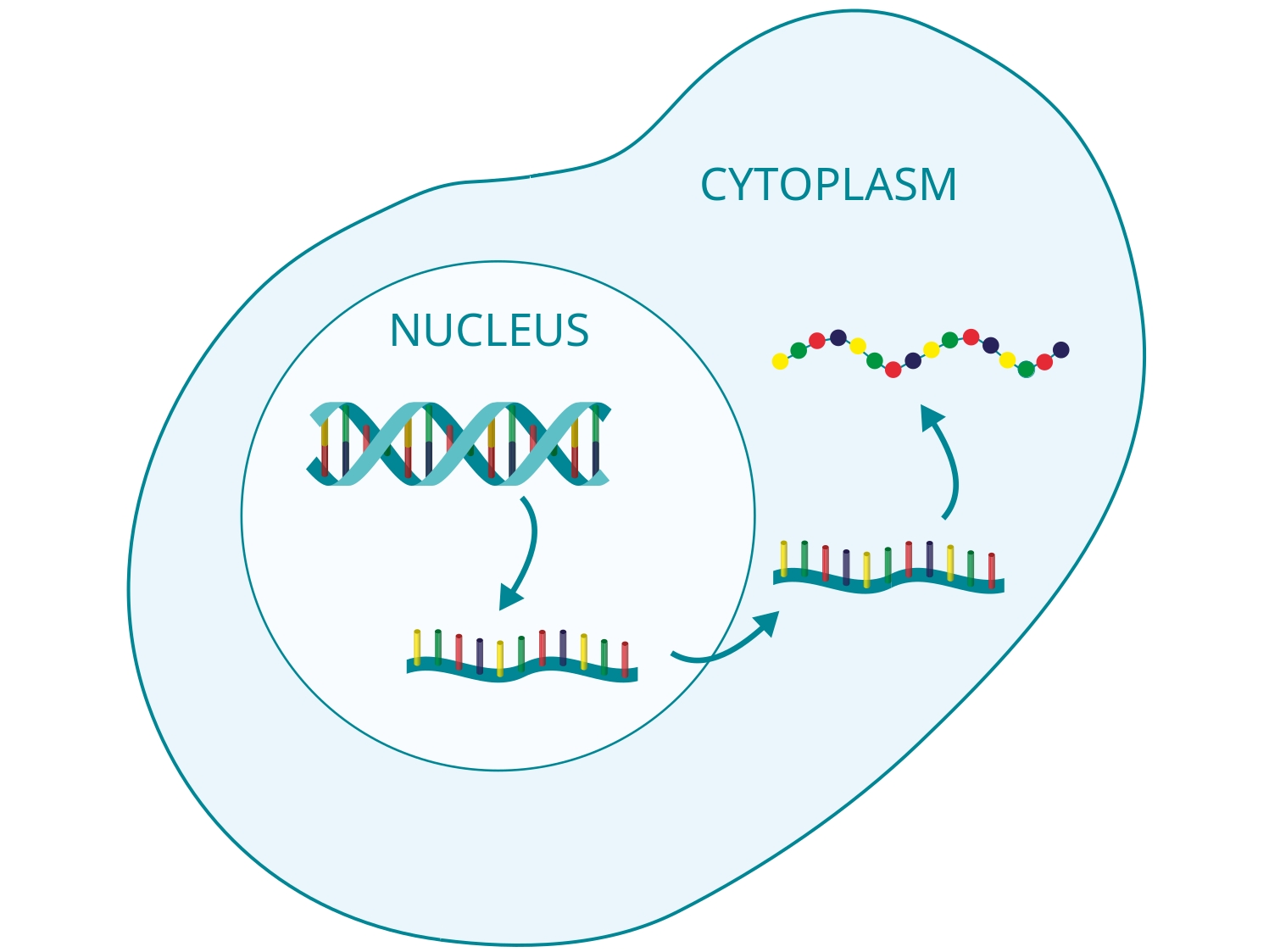
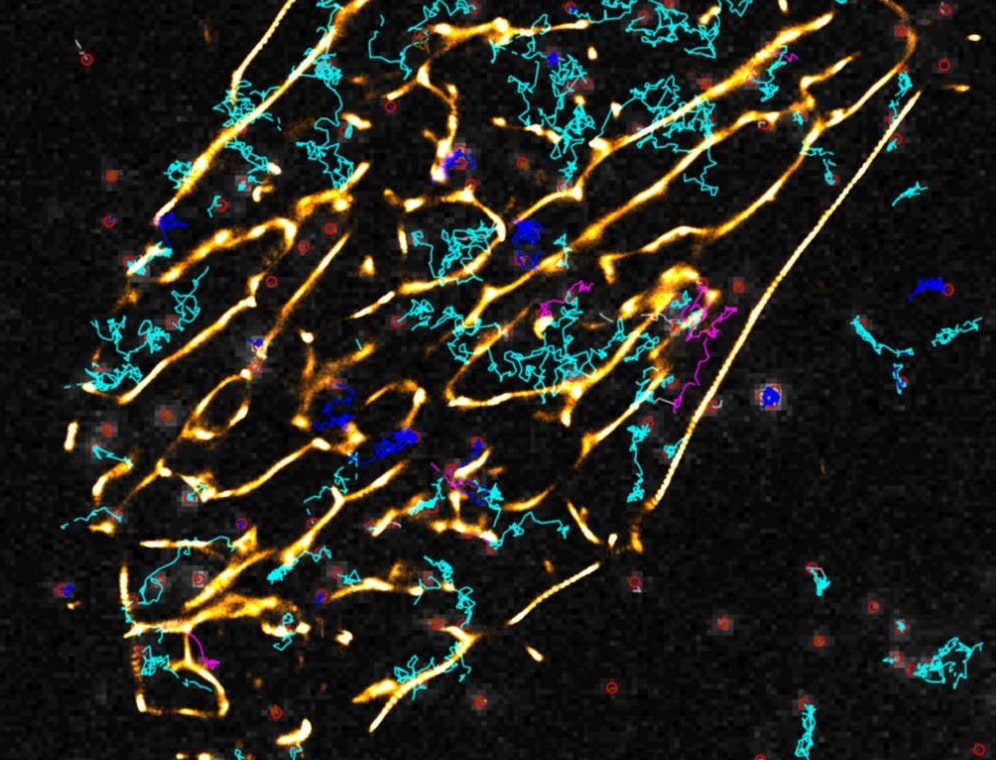
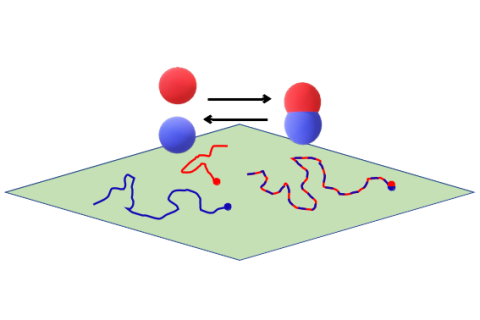
In der Arbeitsgruppe Computational Systems Biology am Zuse Institut Berlin geht es um mathematische Modellierung von biologischen und biochemischen Prozessen. Modellansätze reichen von stochastischen, partikelbasierten Modellen, bei denen die räumliche Bewegung und paarweise Interaktion einzelner Partikel beschrieben werden, bis hin zu deterministischen Systemen ohne räumliche Auflösung. Motiviert durch die Mehrskalenstruktur biologischer Systeme betrachten wir insbesondere hybride Modelle, bei denen die verschiedenen Formalismen kombiniert werden, um so effiziente Algorithmen zur Simulation der dynamischen Systeme zu erhalten. Zu den Anwendungen gehören Kommunikationsvorgänge zwischen Synapsen, Enzymkinetik, oder Genexpression. Über Kooperationen mit Partnern aus den Anwendungsbereichen validieren und verbessern wir unsere Modelle.
Themen
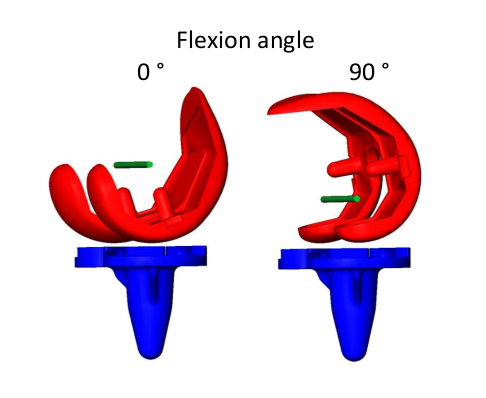
Kinematik künstlicher Knieimplantate
Parametrisierung als Funktion des geometrischen Implantatdesigns